JOH | 我国学者揭示HBV感染宿主后如何自我限制
—— 作者: 时间:2021-09-10
阅读数:
59
编者按:HBV感染威胁着全人类的健康。成人在感染HBV期间,HBV如何在宿主体内自我限制尚不完全清楚。我国学者通过多组学分析,对于HBV与宿主的相互作用、HBV的生命周期进行了系统性的探究,并取得了突破性进展。
2021年,JOH杂志7月刊发表了中国科学院上海生物化学与细胞生物学研究所的一项研究[1],结果提示HBV-宿主相互作用存在着多重自我限制。
研究人员采用基于细胞的HBV复制和基因表达模型,进行RNA测序和核糖体分析,在细胞和小鼠模型中进行蛋白水平的功能评估,并通过基因测序对处于免疫耐受或激活阶段的患者进行HBV亚群的准物种分析。
结果表明,增强子I-stem环(EnhI-SL)是HBV基因组中一个新的顺式元件,发生在EnhI SL的突变可提高病毒聚合酶的表达。研究者发现了一种尚未被充分研究的HBV聚合酶亚型HpZ/P'和一种新的HBX短亚型HBxZ,它们对于HBV的基因表达和基因组复制存在抑制作用。
进一步的研究发现,HBV通过下调宿主RNA剪接体蛋白SRSF2, 可促进病毒前基因组RNA的剪接和HpZ/P'产生,从而抑制HBV cccDNA的转录。SRSF2-HpZ/P'似乎构成了HBV生命周期中的另一种负反馈机制。该项研究提示,靶向宿主的剪接机制可能是一种干预HBV与宿主相互作用的策略。
在此项研究的基础上,北京大学基础医学院鲁凤民教授团队通过长读测序(LRS)技术Iso-Seq研究HBV的整体转录组,为HpZ/P'和HBxZ在转录水平上的表达提供分子生物学证据。该项研究于2021年7月28日在JOH杂志在线发表[2]。
研究人员将prccDNA/pCMV-Cre质粒共转染到Huh-7细胞,然后从上述瞬时转染的细胞中收集总RNA,并按Iso-seq进行测序。结果从4.67 Gbp测序数据中总共获得1059497个全长读取序列,长度范围为531到26451 bp(见图1A-1B)。
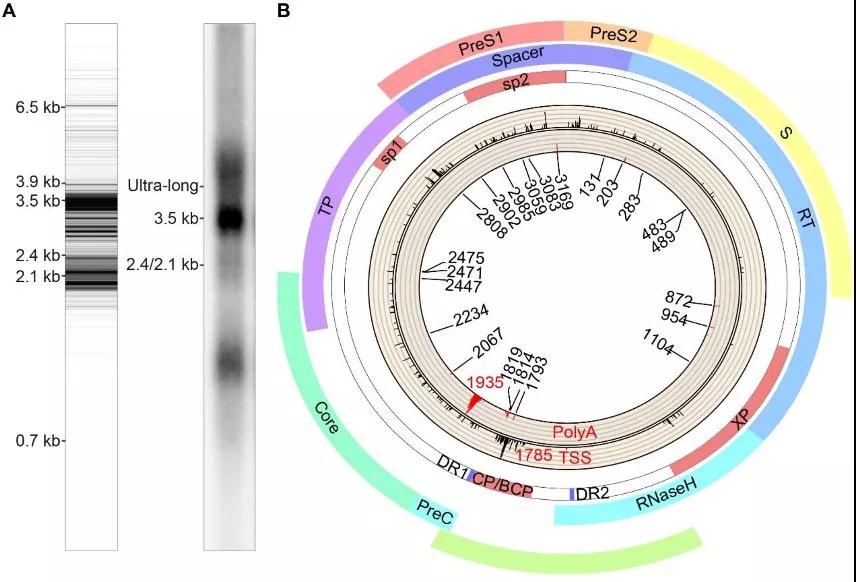
图1.乙型肝炎病毒转录组特征分析
A)HBV转录本的长度分布;B)HBV基因组中TSS(黑色直方图)、TES(红色直方图)和HBV转录本的主要剪接位点(最内层圆圈)的分布
最终获得了12535个与HBV相关的全长读取序列,长度范围为1140到12931 bp。除了经典的3.5 kb、2.4 kb和2.1 kb HBV转录本外,还检测到大量超长HBV转录本。有趣的是,并没有检测到X蛋白的0.7 kb HBx转录本(图1A,左)。在这些超长HBV转录本中,3.9 kb和6.5 kb转录本是主要的亚型。这种超长HBV转录本通过Northern印迹分析进一步验证(图1A,右)。3.9 kb的转录本包含两份HBx 开放阅读框(ORF),转录起始位点(TSS)位于1297-1571 nt。
既往研究报道,HBV感染后0.7 kb的HBx转录本迅速消失[3],3.9 kb的转录本能够翻译X蛋白[4]。因此,研究者推测,除了X蛋白外,HBxZ蛋白也可能主要从3.9 kb的HBx转录本翻译而来。6.5 kb HBV转录本包含两个完整的HBV基因组拷贝,命名为6.5 kb PreC mRNA,因为TSS集中在1785 nt(PreC mRNA的经典TSS)周围。
研究人员进一步分析了本研究中获得的HBV转录本的TSS和转录终止位点(TES)变体。鉴于非经典HBV TESs与HBV选择性剪接位点高度重叠,研究者推测HBV的这些非经典TESs可能是由选择性终止子引起的,即证实了存在基因组RNA选择性剪接事件。研究发现2067 nt、2447 nt、2902 nt、2985 nt、3083 nt、3169 nt、203 nt、283 nt和489 nt是HBV的主要剪接位点。这些位点之间的多重组合构成了HBV的主要剪接转录亚型(图2)。
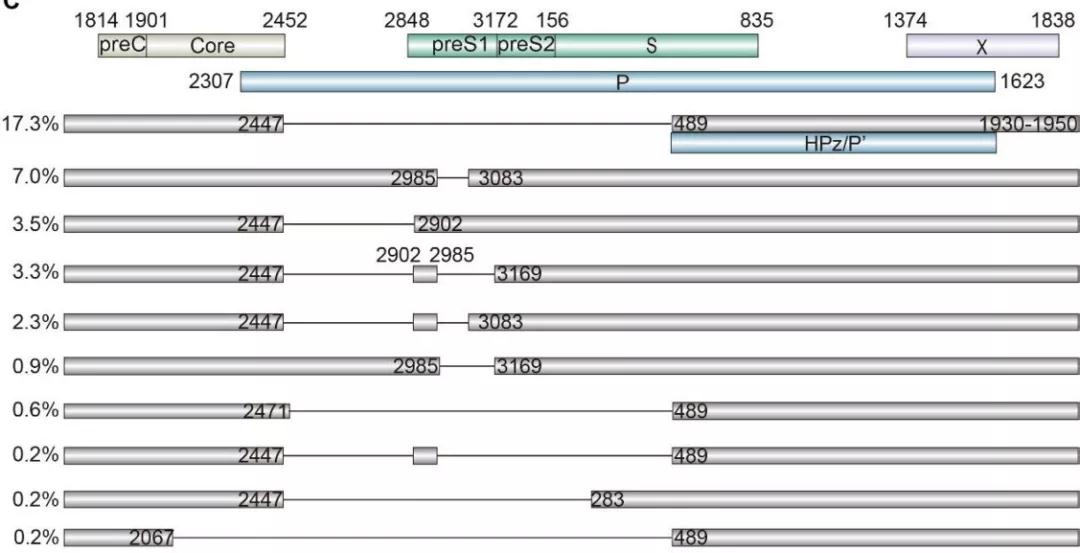
图2.HBV的主要剪接转录亚型
与中国科学院上海生物化学与细胞生物学研究所研究所报道的一致,2447/489是主要的HBV剪接变体,占本研究剪接转录本的17.3%(图2)。在这2447/489个剪接变异体中,高达97.8%是在1682 nt和1814 nt之间,在PreC蛋白的起始密码子之前用TSS转录的。除了PreC、Core和Polymerase蛋白的翻译起始位点外,在由2447/489剪接变异体形成的2446A2447T489G之前还存在多个ATG。因此,与HpZ/P’翻译相关的机制还有待进一步证实。
参考文献:
1.Yuan S, Liao G, Zhang M, et al. Translatomic profiling reveals novel self-restricting virus-host interactions during HBV infection. J Hepatol. 2021 Jul;75(1):74-85.
2.Guan G, Zou J, Zhang T, et al. A global survey of alternative splicing of HBV transcriptome using long-read sequencing. J Hepatol. 2021 Jul 28. Epub ahead of print.
3.Wu HL,Chen PJ,Lin MH,et al. Temporal aspects of major viral transcript expression in Hep G2 cells transfected with cloned hepatitis B virus DNA: with emphasis on the X transcript. Virology. 1991; 185: 644-651
4.Doitsh G,Shaul Y. A long HBV transcript encoding pX is inefficiently exported from the nucleus. Virology. 2003; 309: 339-349
标签:
研究
乙肝
发表评论
全部评论
